确立优势:氮添加显著提升了赖草的重要值,使其在群落中占据主导地位。
促进形态:赖草的株高、茎粗、叶宽等地上部分形态指标均显著增加。
增加生物量:地上部分鲜重和干重随氮梯度的升高而显著增加。
发达根系:总根长、根表面积、根体积等地下根系指标也得到显著提高。
氮添加对禾本科植物赖草(L. secalinus)具有显著的生长促进效应。与其他物种相比,仅有赖草的重要值随着氮添加梯度的升高而显著增加,尤其是在N3和N5处理下。观测发现,与对照组(CK)相比,氮添加显著增加了赖草的株高、茎粗、叶宽及地上生物量。此外,氮添加也显著影响了赖草的根系结构,其总根长、根投影面积、根表面积、根体积、平均根直径和根尖数均受氮添加影响且显著高于对照组 (Fig. 1)。
图1. 氮添加对赖草生长的影响。 (A) 高寒草地物种的重要值。(B) 赖草的株高。(C) 赖草的茎粗。(D) 赖草的叶宽。(E) 单株赖草的地上鲜重。(F) 单株赖草的地上干重。数据为平均值±标准误(n = 3个混合样本,每个含10株植物)。不同字母表示处理间存在显著差异(p < 0.05),由LSD检验得出。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
3.2 氮添加下赖草的碳氮代谢
色素增加:叶绿素a、叶绿素b、总叶绿素和类胡萝卜素含量随氮添加而增加。
光合增强:净光合速率、气孔导度、ΦPSII、ETR和qP等光合参数显著提升。
结构优化:叶绿体膜边界更清晰,类囊体结构更规整,嗜锇颗粒减少。
酶活提高:碳代谢(SS, SPS)和氮代谢(NR, NiR, GOGAT, GDH)相关酶活性被诱导。
元素富集:叶片C、N含量显著增加,C/N比显著下降。
产物重配:可溶性糖和淀粉含量下降,而葡萄糖和总游离氨基酸含量显著增加。
氮添加显著影响了赖草叶片的光合色素、光合参数及叶绿素荧光。叶片中叶绿素a(Chla)、叶绿素b(Chlb)、总叶绿素(Chl)和类胡萝卜素(Car)的含量随氮添加量的增加而显著升高。与对照组相比,氮添加显著增强了叶片的净光合速率、气孔导度、ΦPSII、ETR和qP。此外,高氮处理(N3和N5)显著提高了叶片的Fv / Fm。叶绿体超微结构观察显示,高氮处理下的叶绿体膜边界更清晰完整,类囊体结构排列更有序,嗜锇颗粒数量减少。在代谢层面,高氮处理不仅提高了碳代谢相关酶(SS和SPS)的活性,也诱导了氮代谢相关酶(NR, NiR, GOGAT和GDH)的活性。相应地,叶片C和N含量显著增加,C/N比显著降低。有趣的是,尽管碳代谢相关酶活性升高,但叶片中可溶性糖和淀粉含量却显著降低,仅葡萄糖含量在高氮处理下增加。同时,氮添加显著提高了总游离氨基酸含量并改变了其组分比例 (Fig. 2, Fig. 3)。
图2. 氮添加对赖草光合相关参数的影响。 (A) 叶绿素A。(B) 叶绿素B。(C) 总叶绿素。(D) 类胡萝卜素。(E) 净光合速率。(F) 光系统II的最大光化学效率 (Fv / Fm)。(G) 光系统II的量子产率 (ΦPSⅡ)。(H) 电子传递速率 (ETR)。(I) 光化学猝灭系数 (qP)。(J) 非光化学猝灭 (NPQ)。A–D数据为平均值±标准误(n = 6个混合样本,每个含10株植物),E为(n = 27株植物),F–J为(n = 12株植物)。不同字母表示处理间存在显著差异(p < 0.05),由LSD检验得出。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
图3. 氮添加下赖草叶片中碳氮代谢相关酶活性及产物含量。 (A) 蔗糖合酶 (SS) 活性。(B) 蔗糖磷酸合酶 (SPS) 活性。(C) 硝酸还原酶 (NR) 活性。(D) 亚硝酸还原酶 (NiR) 活性。(E) 谷氨酰胺合成酶 (GS) 活性。(F) 谷氨酸合成酶 (GOGAT) 活性。(G) 谷氨酸脱氢酶 (GDH) 活性。(H) C含量。(I) N含量。(J) C/N比。(K) 可溶性糖含量。(L) 淀粉含量。(M) 葡萄糖含量。(N) 总可溶性游离氨基酸含量及各氨基酸相对含量百分比。左Y轴表示单个游离氨基酸的相对百分比含量,右Y轴表示叶片总游离氨基酸含量。数据为平均值±标准误(n = 3个混合样本,每个含10株植物)。不同字母表示处理间存在显著差异(p < 0.05),由LSD检验得出。*表示各氮沉降处理组与CK组相比,在相对氨基酸含量上存在显著差异。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
3.3 氮添加下赖草的转录响应
基因响应:差异表达基因(DEGs)数量随氮浓度增加而增多。
处理分异:高氮处理(N3, N5)与低氮(CK, N1)在转录组水平上明显分离。
通路富集:DEGs主要富集于光合作用、碳氮代谢及氨基酸代谢等通路。
核心通路:高氮共有的DEGs主要涉及光合、糖代谢、氨基酸代谢和抗性通路。
赖草叶片的转录组对氮添加表现出强烈响应。与对照组相比,N1、N3和N5处理分别鉴定出6985、11435和18522个差异表达基因(DEGs)。PCA分析显示,N3和N5处理组与CK和N1处理组在转录本水平上明显分离。KEGG富集分析表明,所有DEGs主要富集在与碳氮代谢相关的通路,如‘光合作用-天线蛋白’、‘甘氨酸、丝氨酸和苏氨酸代谢’、‘光合作用’、‘光合生物中的碳固定’、‘氮代谢’以及‘淀粉和蔗糖代谢’等。对N3和N5处理共有的DEGs进行分析,发现它们主要参与了与碳代谢相关的‘光合作用-天线蛋白’、‘糖酵解/糖异生’、‘淀粉和蔗糖代谢’通路,与氮代谢相关的‘酪氨酸代谢’、‘甘氨酸、丝氨酸和苏氨酸代谢’通路,以及与胁迫抗性相关的‘植物-病原体互作’通路 (Fig. 4)。
图4. 氮添加下赖草的全局转录组响应。 (A) 不同氮添加处理下的DEGs数量。(B) 各处理组中上调和下调DEGs的韦恩图。(C) 总DEGs的KEGG富集分析前20名。(D) N3和N5处理下共有DEGs的KEGG富集分析前20名(每个条形图上的数字代表DEGs的数量)。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
3.4 氮添加下赖草的蛋白质组动态
蛋白响应:差异表达蛋白(DEPs)数量随氮浓度增加而变化。
功能定位:DEPs主要定位于细胞质和叶绿体,参与碳水化合物、氨基酸和能量代谢。
通路激活:高氮处理激活了光刺激响应、光合作用和光捕获等生物学过程。
核心通路:高氮共有的DEPs主要富集于光合作用、光合天线蛋白和赖氨酸生物合成等。
为补充转录组分析,对氮添加下的赖草叶片进行了蛋白质组学研究。与对照组相比,N1、N3和N5处理中分别有1080、3214和3858个差异表达蛋白(DEPs)。KEGG注释表明,‘碳水化合物代谢’、‘氨基酸代谢’和‘能量代谢’通路包含的DEPs数量最多,且这些DEPs主要定位于细胞质和叶绿体。与转录组学结果相似,N3和N5处理下的DEPs在GO和KEGG通路上更为相似,主要富集于与光合作用相关的通路。对N3和N5处理共有的DEPs进行KEGG富集分析,同样显示它们主要富集在‘光合作用’、‘光合作用-天线蛋白’和‘赖氨酸生物合成’等通路。GO富集分析的圈图显示,与N1处理下多数生物过程被抑制相反,N3和N5处理激活了部分生物过程,包括‘对光刺激的响应’、‘对辐射的响应’以及‘光合作用,光系统I中的光捕获’等 (Fig. 5)。
图5. 氮添加响应下赖草的蛋白质组学变化。 (A) 不同氮添加处理下的DEPs数量。(B) 总DEPs的KEGG功能注释。(C) 总DEPs的亚细胞定位。(D) 各处理组中上调和下调DEPs的韦恩图。(E) N3和N5处理下共有DEPs的KEGG富集分析前20名(每个条形图上的数字代表DEPs的数量)。(F–H) N1、N3和N5处理中DEPs的GO富集分析中生物学过程显著富集的前15个通路的富集圈图。上调和下调的蛋白质分别用红色和蓝色表示,内圈从紫色变为红色表示该生物过程可能被抑制或激活。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
3.5 氮添加下赖草代谢物的积累模式
代谢物谱:共鉴定出2322种代谢物,以脂质、苯丙烷类和聚酮类为主。
处理分异:高氮处理(N3, N5)的代谢谱与对照组显著不同。
差异代谢:差异代谢物(DEMs)数量随氮处理而变化,高氮处理下尤为显著。
通路富集:DEMs主要富集于氨基酸代谢相关通路,如D-氨基酸代谢、酪氨酸代谢等。
核心代谢:谷氨酰胺、天冬氨酸和组氨酸是响应氮沉降的关键代谢节点。
为将转录组和蛋白质组分析与代谢通路联系起来,研究了氮添加下赖草叶片中代谢物的积累变化。共鉴定出2322种代谢物,主要为脂质及类脂分子、苯丙烷类和聚酮类、有机氧化物。与转录组和蛋白质组的PCA分析结果一致,N3和N5处理的样本在代谢组学上也与CK和N1处理显著偏离。与对照组相比,N1、N3和N5处理下分别有360、619和715个差异代谢物(DEMs)。对所有DEMs的富集分析显示,它们主要富集在与氨基酸代谢相关的通路,如‘D-氨基酸代谢’、‘酪氨酸代谢’和‘色氨酸代谢’等。对N3和N5处理共有的DEMs进行KEGG富集分析,同样表明它们主要富集在氨基酸代谢相关通路。KEGG网络图谱进一步证明,所有DEMs相互作用以协调多个代谢通路,其中谷氨酰胺、天冬氨酸和组氨酸是连接最多代谢通路的关键节点,表明这些代谢物在赖草响应氮沉降中起着核心作用 (Fig. 6)。
图6. 氮添加下赖草中代谢物的差异分析。 (A) 所有代谢物的分类饼图。(B) 处理间所有代谢物的PCA分析。(C) 不同氮添加处理下的DEMs数量。(D) 处理组间的UpSet韦恩图。(E) 所有DEMs的KEGG富集分析前20名。点的大小代表DEMs的数量,蓝色越深代表p值越低。(F) N1、N3和N5处理中DEMs的KEGG通路差异丰度得分图。X轴表示差异丰度得分(DA Score),Y轴表示KEGG代谢通路名称。DA得分反映了代谢通路中所有代谢物的总体变化,线段长度表示DA得分的绝对值。点的大小表示通路中注释到的DEMs数量。分布在中心轴右侧且线段较长的点表示通路整体表达趋于上调,反之则趋于下调。* p < 0.05, ** p < 0.01 或 *** p < 0.001。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
3.6 氮添加下赖草的调控通路
光合上调:光系统I、II、天线蛋白、细胞色素b6f复合物等基因和蛋白普遍上调。
碳流加速:碳固定、糖酵解及淀粉蔗糖代谢通路中多数基因和蛋白表达上调。
碳物再配:尽管碳代谢加速,但碳水化合物含量下降。
氮流增强:氮同化、TCA循环及氨基酸合成通路中的关键基因和蛋白表达上调。
氨基酸增:多数氨基酸及其代谢中间体在高氮处理下显著积累。
抗性激活:应激响应基因、抗逆转录因子、抗氧化酶及抗性激素水平均显著上调。
鉴于转录组和蛋白质组分析均显著富集了与光合作用相关的通路,对编码光合器官的DEGs和DEPs进行了特异性分析。结果显示,编码光系统I蛋白、光系统II蛋白、天线蛋白、细胞色素b6f复合物蛋白、铁氧还蛋白、光合电子传递蛋白及叶绿体ATP合酶蛋白的大多数转录本和蛋白质,在氮添加下均被上调,尤其是在N3和N5处理中。同时,与碳水化合物代谢相关的通路,如‘光合生物中的碳固定’、‘糖酵解/糖异生’和‘淀粉和蔗糖代谢’,其相关基因和蛋白的表达水平也显著上调,表明氮添加促进了赖草叶片中的碳代谢过程。然而,代谢物分析显示,尽管碳代谢加速,叶片中的碳水化合物含量在N3和N5处理下却有所下降。在氮代谢方面,编码NIR、GS、GOGAT和GDH的基因或蛋白表达显著上调,增强了氮同化能力。同时,TCA循环及氨基酸合成通路中关键基因和蛋白的表达也得到增强,促进了相关氨基酸及其代谢中间体的积累。最后,与环境适应性相关的基因、转录因子(NAC, bZIP, bHLH等)、抗氧化酶(SOD, POD, APX等)以及抗性相关植物激素(脱落酸和茉莉酸甲酯)的水平,在氮添加下均显著上调,尤其是在N3和N5处理中 (Fig. 7, Fig. 8, Fig. 9, Fig. 10)。
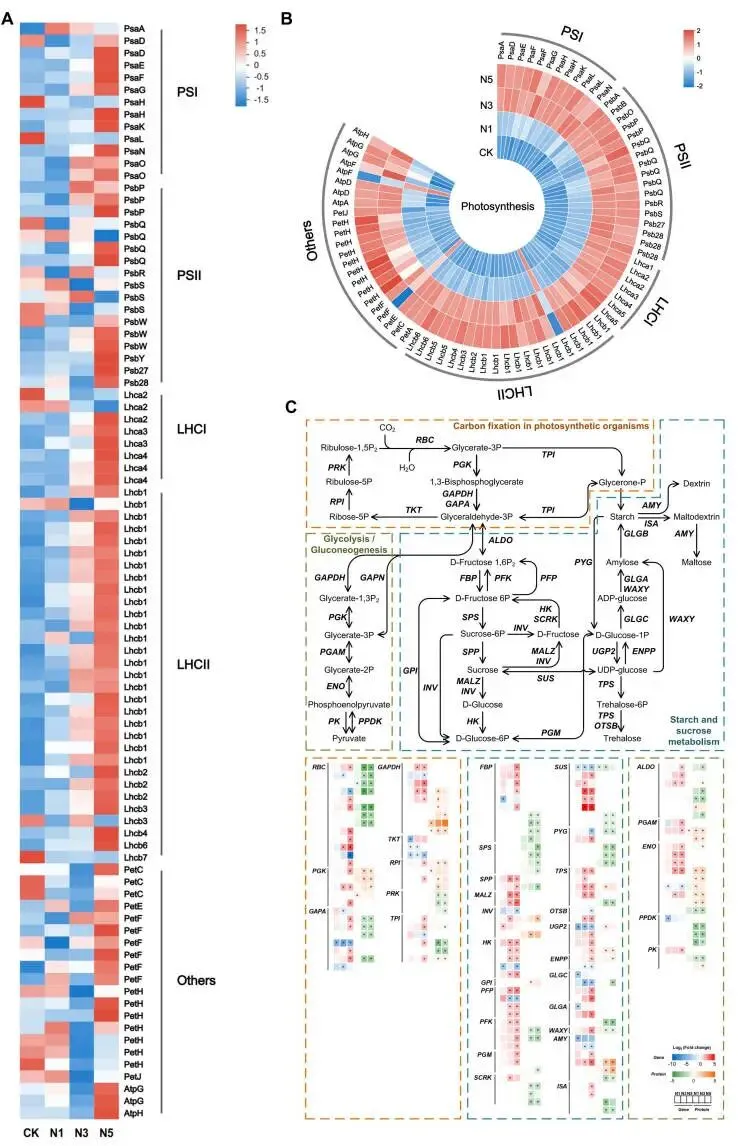
图7. 氮添加下赖草中与光合作用和碳代谢过程相关的基因和蛋白的调控。 (A) 参与光合作用的转录本表达谱。热图颜色表示基因表达的高(红)或低(蓝)。(B) 参与光合作用的蛋白质表达谱。热图颜色表示蛋白质表达的高(红)或低(蓝)。(C) 参与碳代谢过程的基因和蛋白质表达谱。标度尺使用各处理相对于CK的log2(倍数变化)值创建。红色或蓝色表示基因的log2(倍数变化)值。橙色或绿色表示蛋白质的log2(倍数变化)值。ALDO, 果糖二磷酸醛缩酶; AMY, 淀粉酶; ENO, 烯醇化酶; ENPP, 外切核苷酸焦磷酸酶/磷酸二酯酶; FBP, 果糖-1,6-二磷酸酶I; GAPA, 甘油醛-3-磷酸脱氢酶(NADP⁺)(磷酸化); GAPDH, 甘油醛-3-磷酸脱氢酶(磷酸化); GAPN, 甘油醛-3-磷酸脱氢酶(NADP⁺); GLGA, 淀粉合酶; GLGB, 1,4-α-葡聚糖分支酶; GLGC, 葡萄糖-1-磷酸腺苷酰转移酶; GPI, 葡萄糖-6-磷酸异构酶; HK, 己糖激酶; INV, β-呋喃果糖苷酶; ISA, 异淀粉酶; MALZ, α-葡萄糖苷酶; OTSB, 海藻糖-6-磷酸磷酸酶; PFK, 6-磷酸果糖激酶1; PFP, 二磷酸依赖性磷酸果糖激酶; PGAM, 2,3-双磷酸甘油酸依赖性磷酸甘油酸变位酶; PGK, 磷酸甘油酸激酶; PGM, 磷酸葡萄糖变位酶; PK, 丙酮酸激酶; PPDK, 丙酮酸,正磷酸双激酶; PRK, 磷酸核酮糖激酶; PYG, 糖原磷酸化酶; RBC, 核酮糖二磷酸羧化酶; RPI, 核糖-5-磷酸异构酶; SCRK, 果糖激酶; SPP, 蔗糖-6-磷酸酶; SPS, 蔗糖-磷酸合酶; SUS, 蔗糖合酶; TKT, 转酮醇酶; TPI, 磷酸丙糖异构酶; TPS, 海藻糖-6-磷酸合酶/磷酸酶; UGP2, UTP-葡萄糖-1-磷酸尿苷酰转移酶; WAXY, 颗粒结合淀粉合酶。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
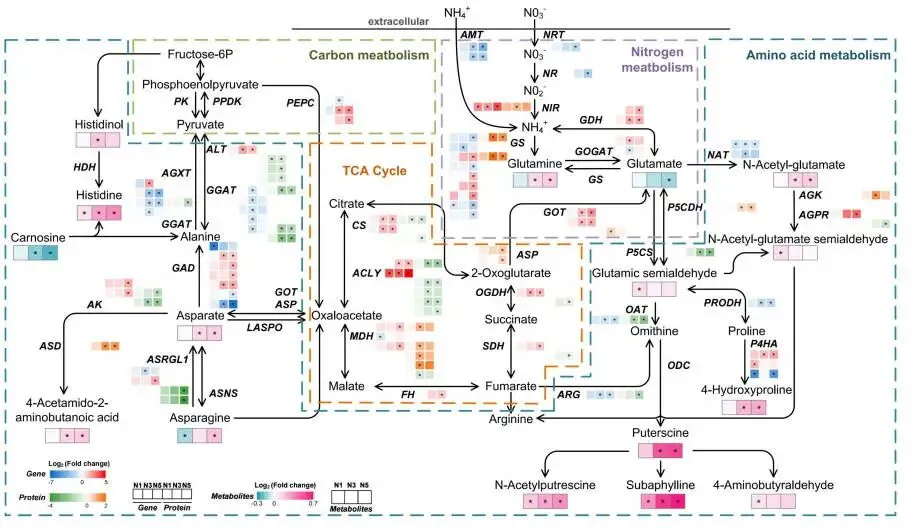
图8. 氮添加下赖草中与氮代谢过程相关的基因和蛋白的调控。 标度尺使用各处理相对于CK的log2(倍数变化)值创建。红色或蓝色表示基因的log2(倍数变化)值。橙色或绿色表示蛋白质的log2(倍数变化)值。粉色或青绿色表示代谢物的log2(倍数变化)值。ACLY, ATP柠檬酸(pro-S)-裂解酶; AGK, 乙酰谷氨酸激酶; AGPR, N-乙酰-γ-谷氨酰-磷酸还原酶; AGXT, 丙氨酸-乙醛酸转氨酶; AK, 天冬氨酸激酶; ALT, 丙氨酸转氨酶; AMT, 铵转运蛋白; ARG, 精氨酸酶; ASD, 天冬氨酸-半醛脱氢酶; ASNS, 天冬酰胺合成酶(谷氨酰胺水解); ASP, 天冬氨酸转氨酶, 叶绿体; ASRGL1, L-天冬酰胺酶/β-天冬氨酰肽酶; CS, 柠檬酸合酶; FH, 延胡索酸水合酶; GAD, 谷氨酸脱羧酶; GDH, 谷氨酸脱氢酶; GGAT, 谷氨酸-乙醛酸转氨酶; GOGAT, 谷氨酸合酶; GOT, 天冬氨酸转氨酶, 线粒体; GS, 谷氨酰胺合成酶; HDH, 组氨醇脱氢酶; LASPO, L-天冬氨酸-天冬氨酸氧化酶; MDH, 苹果酸脱氢酶; NIR, 亚硝酸还原酶; NR, 硝酸还原酶; NRT, 硝酸盐/亚硝酸盐转运蛋白; OAT, 鸟氨酸-酮酸转氨酶; ODC, 鸟氨酸脱羧酶; OGDH, 2-酮戊二酸脱氢酶; P4HA, 脯氨酰4-羟化酶; P5CDH, 1-吡咯啉-5-羧酸脱氢酶; P5CS, δ-1-吡咯啉-5-羧酸合成酶; PEPC, 磷酸烯醇式丙酮酸羧化酶; PK, 丙酮酸激酶; PPDK, 丙酮酸,正磷酸双激酶; PRODH, 脯氨酸脱氢酶; SDH, 琥珀酸脱氢酶。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
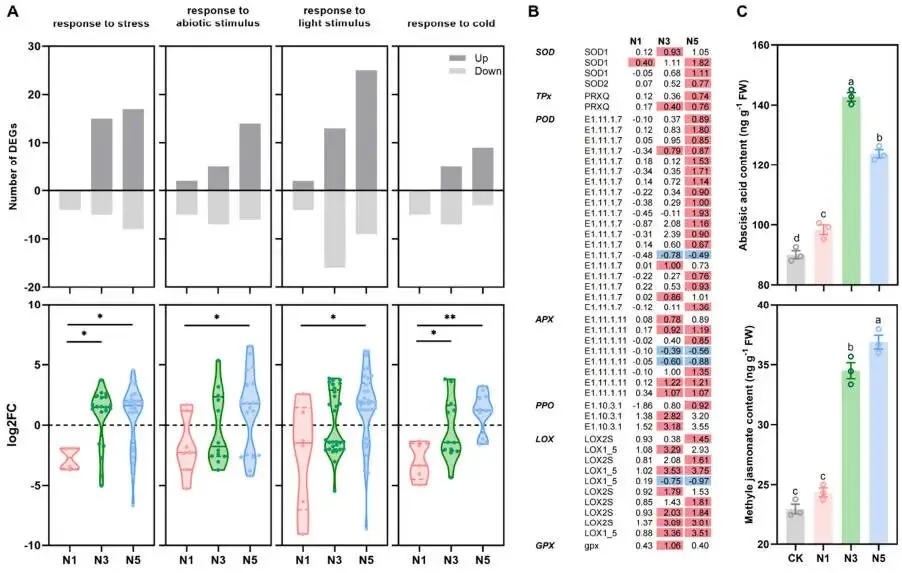
图9. 氮添加下赖草对环境的适应性。 (A) GO术语‘应激响应’、‘非生物刺激响应’、‘光刺激响应’和‘冷响应’中DEGs的表达谱。Y轴分别为上调和下调DEGs的数量以及各处理相对于CK的DEGs的log2(倍数变化)。* p < 0.05, ** p < 0.01 或 *** p < 0.001。(B) 编码抗氧化酶的DEPs,值为各处理相对于CK的log2(倍数变化)。方框颜色代表上调(红)和下调(蓝)的蛋白质。APX, 抗坏血酸过氧化物酶; GPX, 谷胱甘肽过氧化物酶; LOX, 脂氧合酶; POD, 过氧化物酶; PPO, 多酚氧化酶; SOD, 超氧化物歧化酶; TPx, 硫氧还蛋白过氧化物酶。(C) 脱落酸和茉莉酸甲酯含量。数据为平均值±标准误(n = 3个混合样本,每个含10株植物)。不同字母表示处理间存在显著差异(p < 0.05),由LSD检验得出。CK, 0 kg N ha⁻¹ year⁻¹; N1, 8 kg N ha⁻¹ year⁻¹; N3, 40 kg N ha⁻¹ year⁻¹; N5, 72 kg N ha⁻¹ year⁻¹。
图10. 赖草对氮沉降响应机制的概述。 不同箭头类型分别表示促进、不显著或抑制。