本科毕业于山东农业大学,博士毕业于南京农业大学,南京农大教授以通讯作者身份在一区Top期刊上发表研究论文
- 2026-02-09 07:23:23



近日,南京农业大学陈发棣教授团队在《New Phytologist》期刊上发表题为“Sequential Oligo-FISH reveals conserved synteny and rapid cytological diploidization in Chrysanthemum autopolyploids”的研究成果。南京农业大学菊花遗传与种质创新利用团队博士毕业生何俊为论文第一作者,南京农业大学园艺学院王海滨教授和陈发棣教授为论文通讯作者。
研究团队基于已发表的栽培菊花‘钟山紫桂’高质量单倍体基因组,首次开发出菊属全染色体组特异性涂染探针库。此外,团队进一步创新性地建立了顺序荧光原位杂交技术体系,首次实现了对菊属所有同源染色体组的逐条、精准识别与可视化,攻克了长期以来菊属染色体组“分辨难”的核心技术难题。
基于开发的寡核苷酸探针库(CmOP-1及CmOP-2)、染色体特异涂染探针及顺序荧光原位杂交技术体系,团队对7份菊属种质进行顺序荧光原位杂交分析,以携带5S rDNA位点的同源染色体组(Chr 19/20/21)为例证实了菊属在演化过程中保持着极其保守的染色体共线性,未发现大规模易位或重排。这为解析菊属稳定的进化模式提供了直接可视化的细胞学证据。
基于人工创制的同源多倍体材料及顺序FISH技术,进一步研究发现了菊属同源多倍体在形成过程中伴随快速二倍化现象,即多倍体细胞在减数分裂中表现出类似二倍体的二价体配对模式。基于相关Oligo-FISH及减数分裂染色体行为统计结果,研究人员认为:重复序列(尤其是异染色质区富集的重复序列)的动态变化(扩增与丢失),可能通过影响减数分裂过程中同源染色体的识别与配对,从而促进了这种快速二倍化,以维持多倍体基因组的稳定性。
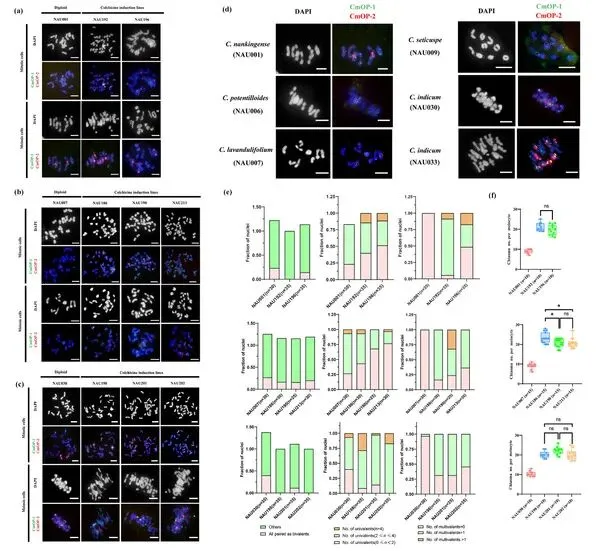
菊属同源多倍体减数分裂染色体行为观察统计

